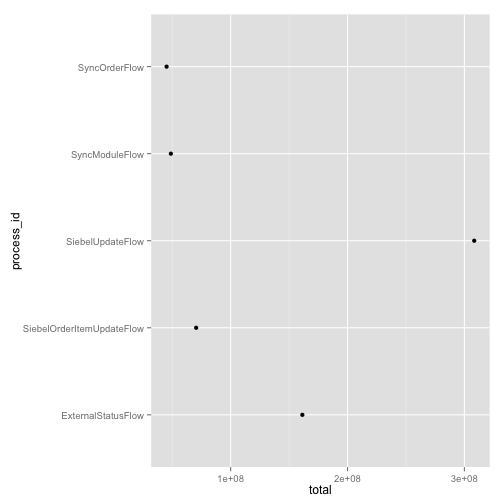
data_db <- tbl(my_db, sql("SELECT * FROM P1"))
data_db
## Source: postgres 8.4.18 [diego@ocos:5432/postgres]
## From: <derived table> [?? x 29]
##
## cikey domain_ref process_id revision_tag
## 1 124642631 0 ExternalStatusFlow 1.0
## 2 124642639 0 SiebelUpdateFlow 1.0
## 3 124642659 0 SiebelUpdateFlow 1.0
## 4 124642663 0 SiebelUpdateFlow 1.0
## 5 124642667 0 SyncOrderFlow 1.0
## 6 124642678 0 SiebelUpdateFlow 1.0
## 7 124642682 0 SiebelUpdateFlow 1.0
## 8 124642685 0 SiebelUpdateFlow 1.0
## 9 124642690 0 SyncOrderFlow 1.0
## 10 124642695 0 ODSOrderSyncFlow 1.0
## .. ... ... ... ...
## Variables not shown: creation_date (time), creator (chr), modify_date
## (time), modifier (chr), state (int), priority (int), title (chr), status
## (chr), stage (chr), conversation_id (chr), root_id (chr), parent_id
## (chr), scope_revision (int), scope_csize (int), scope_usize (int),
## process_guid (chr), process_type (int), metadata (chr), ext_string1
## (chr), ext_string2 (chr), ext_int1 (int), test_run_id (chr), at_count_id
## (int), at_event_id (int), at_detail_id (int)